
张长生研究团队揭示免疫抑制剂浅蓝霉素生物合成的底物回补机制
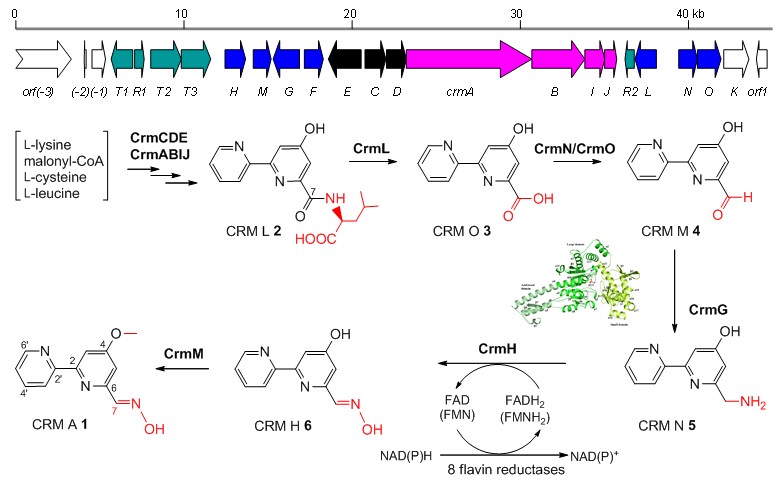
图1. 浅蓝霉素A(CRM A 1)的生物合成基因簇及其生物合成途径
阐明CRM A的生物合成途径是将组合生物合成技术应用于CRM A结构改造和产量优化的前提。因此,研究人员首先从海洋放线菌A. cyanogriseus WH1-2216-6中克隆和鉴定了CRM A的生物合成基因簇,阐明了CRM A中二联吡啶环的骨架结构起源于杂合的聚酮合酶/非核糖体肽合成酶(PKS/NRPS)途径,并发现了一种独特的生物合成策略:通过添加一种冗余的氨基酸来保护生物合成中间体,以形成正确的二联吡啶环的骨架结构,随后通过氨基水解酶切除保护基团(L-亮氨酸),确保生物合成途径往下游进行(Org Lett, 2012, 14, 2666-2669; "Hot off the press" in Nat Prod Rep, 2012, 29, 829-833)。紧接着,研究人员阐明了CRM A生物合成中的CrmG催化的氨基转移过程(ACS Chem Biol, 2016, 11, 943-952)、二元组分单加氧酶CrmH催化的肟基形成机制(J Am Chem Soc, 2013, 135, 18750-18753; "Hot off the press" in Nat Prod Rep, 2014, 31, 414-418)和CrmM催化的甲基化过程(Org lett, 2014, 16,4264-4267),从而完整解析了CRM A生物合成的后修饰过程(图1)。
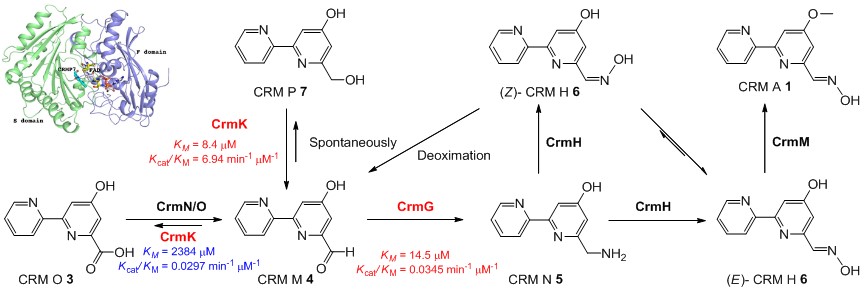
图2. 浅蓝霉素A生物合成中底物补偿机制的发现
氨基转移酶复杂的“乒乓”转氨机制导致CrmG转氨过程非常缓慢(Km 14.5 μM, kcat/Km 0.0345 min-1 μM-1),同时发现CrmH催化产生的产物(Z)-CRM H 6极不稳定,自发脱肟生成醛基,两个方面的原因导致了醛基化合物CRM M 4在细胞内会大量累积。而醛基产物往往会与细胞内其他蛋白的胺和硫醇发生加成反应产生毒性,为消除毒性,醛基会在细胞内被还原成酸或醇。与此理论相吻合,我们发现在CRM A 1的生物合成中过量累积的醛基化合物CRM M 4会通过坎尼扎罗反应(Cannizzaro反应)自发还原成CRM P 7,因此,在crmG突变株中我们发现的累积产物是CRM P 7而不是预想的产物CRM M 4(ACS Chem Biol, 2016, 11, 943-952)。在CRM生物合成基因簇的末端存在一个编码核黄素氧化酶的基因crmK,其突变株仍能生产CRM A 1,表明crmK在浅蓝霉素A的生物合成过程中是非必须基因。意外的是,在crmK突变株中同时发现了副产物CRM P 7。我们通过体外生化实验证实CrmK能够以CRM P 7为底物进行两步连续的氧化反应,顺次生成CRM M 4和CRM O 3。酶动力学研究表明第二步氧化反应CRM M 4至CRM O 3在细胞内很难发生(Km值很大,kcat很小),这样即保证了副产物CRM P可以被重新利用,同时又避免了过度氧化导致能量的浪费(图2)。晶体结构解析显示CrmK采用两个独立的催化位点保证CRM P 7到CRM M 4的转化,为CrmK高效催化醇到醛提供了进一步的理论依据。(Chem Sci, 2016, DOI: 10.1039/C6SC00771F)
在次级代谢产物生物合成基因簇中除了合成化合物骨架的结构基因外,还包括分泌、免疫、调控和基团保护等功能的基因。本研究首次通过遗传、生化和晶体学数据揭示了氧化酶CrmK介导的底物回补机制,为次级代谢产物基因簇中未知功能基因的研究提供了新思路。
这些工作主要由朱义广博士、徐进新博士、付鹏博士和梅显贵等完成,获得了国家自然科学基金委、科技部、中科院和广东省等项目的资助。
相关论文列表:
(1) Zhu, Y#., Picard, M#., Zhang, Q., Barma, J., Despres, X., Mei, X., Zhang, L., Duvignaud, J., Couture, M., Zhu, W., Shi, Rong*., Zhang, C*. Flavoenzyme CrmK-mediated substrate recycling in caerulomycin biosynthesis. Chemical Science. 2016, DOI: 10.1039/C6SC00771F.
(2) Zhu, Y#., Xu, J#., Mei, X#., Feng, Z., Zhang, L., Zhang, Q., Zhang, G., Zhu, W*., Liu, J*., Zhang, C*. Biochemical and structural insights into the aminotransferase CrmG in caerulomycin biosynthesis. ACS Chemical Biology. 2016, 11(4): 943-952.
(3) Fu, P#., Zhu, Y#., Mei, X., Wang, Y., Jia, H., Zhang, C*., Zhu, W*. Acyclic congeners from Actinoalloteichus cyanogriseus provide insights into cyclic bipyridine glycoside formation. Organic Letters. 2014, 16(16): 4264-4267.
(4) Zhu, Y., Zhang, Q., Li, S., Lin, Q., Fu, P., Zhang, G., Zhang, H., Shi, R., Zhu, W*., Zhang, C*. Insights into caerulomycin a biosynthesis: a two-component monooxygenase CrmH-catalyzed oxime formation. Journal of the American Chemical Society. 2013, 135(50): 18750-18753
(5) Zhu, Y#., Fu, P#., Lin, Q., Zhang, G., Zhang, H., Li, S., Ju, J., Zhu, W*., Zhang, C*. Identification of caerulomycin a gene cluster implicates a tailoring amidohydrolase. Organic Letters. 2012, 14(11): 2666-2669.
附件下载: